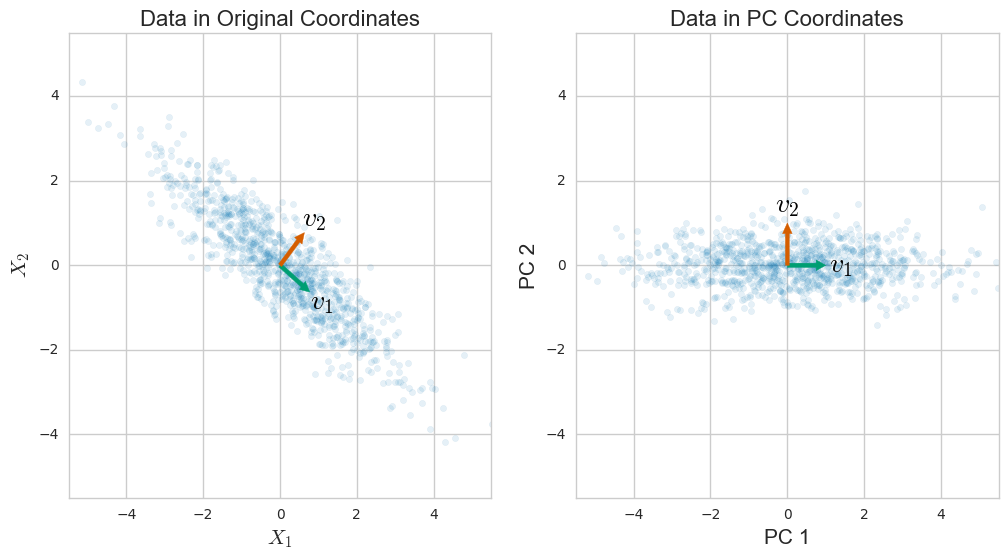
Seja a matriz de dados tamanho de n × p , onde n é o número de amostras ep é o número de variáveis. Vamos supor que ele esteja centralizado , ou seja, os meios das colunas foram subtraídos e agora são iguais a zero.Xn × pnp
Em seguida, o matriz covariância C é dada por C = X ⊤ X / ( n - 1 ) . É uma matriz simétrica e, portanto, pode ser diagonalizada: C = V L V ⊤ , onde V é uma matriz de vetores próprios (cada coluna é um vetor próprio) e L é uma matriz diagonal com valores próprios λ i em ordem decrescente na diagonal . Os autovetores são chamados eixos principais oup×pCC=X⊤X/(n−1)
C=VLV⊤,
VLλi principais direções principaisdos dados. As projeções dos dados nos eixos principais são denominadas
componentes principais , também conhecidas como
pontuação do PC ; elas podem ser vistas como novas variáveis transformadas. O
-ésimo componente principal é dada por
J coluna -ésimo de
X V . As coordenadas do
i ponto de dados -ésimo no novo espaço PC são dadas pelo
i fileira -ésimo de
X V .
jjXViiXV
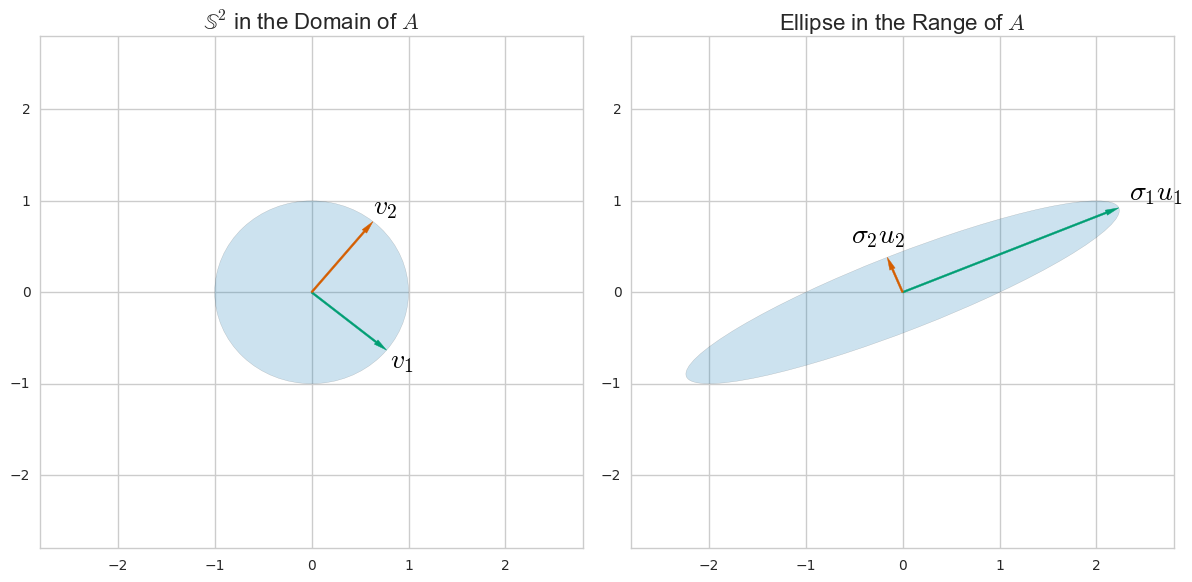
Se agora realizarmos a decomposição do valor singular de , obteremos uma decomposição X = U S V ⊤ , onde U é uma matriz unitária e S é a matriz diagonal dos valores singulares s i . A partir daqui, pode-se ver facilmente que C = V S U ⊤ U S V ⊤ / ( n - 1 ) = V S 2X
X=USV⊤,
USsio que significa que os vectores singulares certas
Vsão direcções principais e que os valores singulares estão relacionados com os valores próprios da matriz covariância por meio de
λi=s 2 i /(n-1). Os componentes principais são dadas por
XV=LSV⊤V=LS.
C=VSU⊤USV⊤/(n−1)=VS2n−1V⊤,
Vλi=s2i/(n−1)XV=USV⊤V=US
Para resumir:
- Se , as colunas de V são as principais direções / eixos.X=USV⊤V
- US
- λi=s2i/(n−1)λi
- n−1−−−−−√UVS/n−1−−−−−√
- XX⊤X/(n−1)
- XUV
- X
- pk<pkUk×kSUkSkn×kk
- kV⊤kXk=U⊤kS⊤kV⊤kn×pkXkk
- Un×nVp×pn>pn−pUSUn×pn≫pUn≪p
Links adicionais